La Red Regional de Vigilancia Genómica de COVID-19 (COVIGEN), coordinada por la OPS y conformada por laboratorios de 24 países de la región, monitorea el virus que causa la COVID-19 para detectar cualquier cambio en su secuencia que pueda influir sobre su capacidad de propagación, la gravedad de la enfermedad, la eficacia de las vacunas, los tratamientos, el diagnóstico u otras medidas de control en salud pública y social. ¿Cómo funciona la red? ¿Cuál es la distribución actual de las variantes de preocupación? Y ¿Por qué es importante esta red para ayudar a poner fin a la pandemia y prepararse para la futura?
— Julio 2021 —
Cincuenta muestras de personas que dieron positivas para COVID-19 parten del aeropuerto de Santo Domingo en República Dominicana hacia Río de Janeiro. El destino final es el laboratorio de la Fundación Oswaldo Cruz (FIOCRUZ) donde se realizará la secuenciación genómica del virus SARS-CoV-2. Los resultados de esas muestras seleccionadas estratégicamente y en forma representativa de las diferentes regiones del país permitirán trazar un panorama de las variantes circulantes y asesorar las medidas de salud pública para hacer frente a la pandemia. La falta de capacidad instalada para hacer secuenciación genómica y vigilar el virus que causa la COVID-19 no es un problema para los países de las Américas desde que en marzo de 2020 la Organización Panamericana de la Salud (OPS) creó la Red Regional de Vigilancia Genómica de COVID-19 (COVIGEN), que integran laboratorios de salud pública de 24 países de la región.
"La secuenciación genómica de los virus es un proceso relativamente nuevo y complejo que requiere de una gran inversión que va desde equipos de alto costo, recursos humanos capacitados, reactivos y otros insumos, y no todos los países del mundo y de la región tienen esa capacidad instalada en sus laboratorios de salud pública para hacerla”, señaló Jairo Méndez Rico, asesor en enfermedades virales emergentes de la OPS.
Sin embargo, agregó, “la Red Regional de Vigilancia Genómica viene a disminuir esa brecha de acceso a la secuenciación para todos los países de la región que lo requieran”.
La vigilancia es efectiva cuando las muestras que se analizan son representativas de lo que está sucediendo en un país y están asociadas a la epidemiología de la enfermedad, a los viajeros, o a algún brote o conglomerado.
Trece países que participan en la red tienen una capacidad limitada o están en proceso de ampliar sus capacidades para hacer secuenciación, por lo que envían sus muestras a alguno de los seis laboratorios regionales de referencia ubicados en Brasil, Chile, Estados Unidos, México, Panamá y Trinidad y Tobago. Estos esfuerzos permitieron, por ejemplo, detectar por primera vez variantes de preocupación en Bolivia, Haití, Honduras, Guatemala y la República Dominicana.
La red, a través de la OPS y del intercambio entre laboratorios participantes, también ha fortalecido a los diez países que sí tenían alguna capacidad de secuenciación. En ese sentido, la OPS ha brindado apoyo mediante protocolos para el tamizaje de las variantes de preocupación, colaboración para la realización de los análisis de los resultados, donación de reactivos y entrenamientos al personal de laboratorio.
La vigilancia del SARS-CoV-2 es clave para detectar cualquier cambio en la secuencia del virus que pueda influir sobre su propagación, la gravedad de la COVID-19 o la eficacia de las vacunas, los tratamientos, el diagnóstico u otras medidas de salud pública y social. “Seguirle los pasos al virus es una medida fundamental para poder informar oportunamente a los países y al público sobre los posibles cambios que puedan generar retos en la respuesta al virus y evitar su propagación”, sostuvo Méndez Rico.
El trabajo de la red también permitió no solo incrementar la cantidad de datos de secuenciación disponibles, sino también la calidad de estos a nivel regional y aportar al conocimiento global sobre el virus. De reportarse inicialmente datos solo de Estados Unidos y Canadá, la región reporta actualmente datos de 49 países. “De esta manera, tenemos una imagen de lo que está sucediendo también en la región y como se van distribuyendo estos virus”, indicó el asesor de la OPS.
“No existe un número específico de muestras a secuenciar por país para vigilar el SARS-CoV-2, pero no se trata de secuenciar por secuenciar”, explicó Méndez Rico. “La vigilancia es efectiva cuando las muestras que se analizan son representativas de lo que está sucediendo en un país y están asociadas a la epidemiología de la enfermedad, a los viajeros, o a algún brote o conglomerado”, agregó.
Hasta el momento, la evidencia disponible ha demostrado que algunas variantes de preocupación tienen mayor capacidad para transmitirse y replicarse, sin embargo, no hay evidencia suficiente para afirmar que son más agresivas o severas, y su impacto en las reinfecciones o escape de la respuesta inmune aún debe ser establecido.
Dar a conocer una secuencia del virus que causa la COVID-19 inmediatamente a nivel global a través de la Iniciativa Global para Compartir Datos de Influenza, conocida como GISAID por sus siglas en inglés, también es clave para que se tomen acciones de salud pública, se mejoren protocolos de diagnóstico o se genere información para el desarrollo de vacunas. “Todos -tanto los laboratorios públicos como los privados- debemos aportar al conocimiento de la COVID-19 y a su respuesta”, subrayó.
Para el asesor en enfermedades virales emergentes de la OPS, la Red Regional de Vigilancia Genómica ha plantado la semilla “para que la vigilancia genómica se haga en forma rutinaria en las Américas cuando se termine la pandemia”, algo que puede contribuir no solo a la vigilancia de otros virus ya circulantes, como los del dengue, los arenavirus y los de influenza, sino también para detectar nuevos virus o patógenos que pueden surgir y estar así más y mejor preparados para enfrentar la próxima pandemia.
Variantes de interés y preocupación en las Américas
La mutación es parte natural del proceso de evolución de los virus. Desde su caracterización genómica inicial, el SARS-CoV-2 ha mutado y evolucionado. Un virus mutado se considera una variante del virus original, y aunque la mayoría de las mutaciones no tiene ningún impacto, algunas pueden ser ventajosas para el virus y resultar en que se vuelva más transmisible o facilitar que se escape de la respuesta inmune, ya sea natural si la persona estuvo infectada o la generada por la vacuna.
La OMS ha designado un grupo de variantes de interés y de preocupación, que se actualiza en función de la aparición e identificación de nuevas variantes que tengan impacto significativo y demostrado en la salud pública global. Las autoridades de los países pueden también designar otras variantes de interés o preocupación local.
Una variante es considerada de interés (VOI) cuando ha cambiado en relación con el virus original y se ha identificado que causa transmisión comunitaria/múltiples casos/clústeres de COVID-19, o se ha detectado en varios países. Hasta julio de 2021, la OMS identificó cuatro variantes de interés a nivel global (eta, iota, kappa y lambda), al menos 2 de las cuales han sido detectadas en las Américas (lambda y Iota).
Variantes de preocupación en las Américas – Hasta el 28 de julio de 2021
Una VOI pasa a ser una variante de preocupación (VOC) si se ha demostrado que está asociada con un aumento de la transmisibilidad o cambio perjudicial en la epidemiología de la COVID-19; si aumenta la virulencia o cambia la presentación clínica de la enfermedad; o si disminuye la eficacia de las medidas de control o del diagnóstico disponible, las vacunas y terapias. Hasta julio de 2021, la OMS identificó cuatro variantes de preocupación a nivel global: alpha, beta, gamma y delta.
La Red Regional de Vigilancia Genómica ha podido identificar alguna de las variantes de preocupación en 49 países de las Américas. Alpha fue detectada en 45 países, beta en 20, gamma en 31 y delta en 20. Todas las cuatro VOC han sido identificadas en 12 países de la región.
"Hasta el momento, la evidencia disponible ha demostrado que algunas variantes de preocupación tienen mayor capacidad para transmitirse y replicarse, sin embargo, no hay evidencia suficiente para afirmar que son más agresivas o severas, y su impacto en las reinfecciones o escape de la respuesta inmune aún debe ser establecido”, sostuvo Méndez Rico.
Asimismo, indicó: “la información muestra que las vacunas existentes protegen contra todas las variantes que circulan actualmente a nivel global”.
En términos prácticos, señaló el experto de la OPS, sigue siendo el mismo virus y las medidas a tomar para prevenir su propagación son las mismas: la vacunación, el distanciamiento físico, el uso de mascarillas y evitar las aglomeraciones, así como el control de las infecciones mediante la realización de pruebas, el rastreo de contactos, aislamientos y las cuarentenas.
Méndez Rico explicó que los virus son organismos que evolucionan. Dijo que los virus necesitan de las células de las personas para multiplicarse. “En su proceso de evolución busca transmitirse más fácilmente a más personas, sin embargo, al virus no le conviene matar a la persona, porque entonces ¿dónde se va a multiplicar?” El especialista de la OPS sostuvo que, en general, los virus evolucionan para convertirse en más transmisibles, pero no necesariamente más agresivos, letales o severos, algo que se presume puede ocurrir también con el virus que causa la COVID-19.
Ministerio de Salud de Paraguay realiza por primera vez secuenciación de SARS-COV
Conocer la secuencia genética del virus que causa la COVID-19 para monitorear o detectar sus variantes no es tarea sencilla, pero ahora el Ministerio de Salud de Paraguay puede hacerlo en su territorio.
La OPS y la Red Regional de Vigilancia Genómica han apoyado al país y fortalecido la capacidad del Laboratorio Central de Salud Pública de Paraguay, que ha comenzado a generar sus propias secuencias del virus SARS-CoV-2.
El Ministerio contaba con uno de los dos equipos disponibles en el país para secuenciar, pero eso no era suficiente. Necesitaba crear capacidad en los técnicos para llevar adelante el proceso y, también, acceder a reactivos. Así, en abril último la OPS movilizó a una especialista del laboratorio de Paraguay hacia Fiocruz, uno de los laboratorios de referencia de la red ubicado en Río de Janeiro, donde fue capacitada durante una semana.
La bioquímica Sandra González fue entrenada en el montaje de la librería genómica, el proceso de secuenciación y en análisis bioinformático para extraer las informaciones de linajes y variantes. Al regresar, junto con el resto del equipo del laboratorio corrieron varias pruebas, y, tras la llegada de todos los materiales necesarios, realizaron con éxito su primera secuenciación a fines de julio pasado.
Vamos a empezar a formar más gente, a transferir capacidad.
La OPS facilitó insumos y reactivos para la realización de las pruebas, algo difícil de conseguir por la alta demanda global y las restricciones de vuelos. El protocolo desarrollado y validado por Fiocruz para realizar la secuenciación también fue compartido para su implementación en el país, y el Instituto Gorgas de Panamá, otro miembro de la red, apoyó al país a la hora de hacer el análisis bioinformático de los resultados.
“Hacer secuenciación es un salto muy importante que nos permitirá optimizar el tiempo para obtener información fundamental y en forma oportuna”, sostiene Cynthia Vázquez, directora técnica del Laboratorio Central de Salud Pública de Paraguay. “El apoyo de la OPS y de los colegas de la Red fue sumamente importante”, agrega.
Cynthia detalla que seguirán utilizando las pruebas PCR en tiempo real para hacer tamizaje de variantes, algo que puede identificar con cierta probabilidad algunas de las variantes de preocupación, y que enfocarán la secuenciación en identificar la variante de preocupación delta y la de interés lambda. Además, cuenta, “vamos a empezar a formar más gente, a transferir capacidad”. Un logro para Paraguay y para los esfuerzos globales contra la pandemia.
Haití fortalece la detección de variantes
Cuando en mayo de 2021 Haití comenzó a registrar un incremento en los casos de COVID-19 inusual para el país hasta ese momento, la necesidad de conocer las variantes circulantes se convirtió en una prioridad.
"Desde que detectamos por primera vez COVID-19 no sabíamos exactamente qué variante era, pero ahora sabemos que se trata de alpha y gamma", afirma Ito Journel, coordinador responsable por el servicio de biología molecular del Laboratorio Nacional de Salud Pública de Haití, sobre las variantes de preocupación detectadas en el país hasta mediados de julio de 2021.
Ito cuenta que la OPS y la Red Regional de Vigilancia Genómica de COVID-19 (COVIGEN) hicieron posible la llegada de muestras al laboratorio de Fiocruz, en Río de Janeiro, Brasil, uno de los seis de referencia regional para secuenciación de COVIGEN que realizan secuenciación para los países de la red que carecen de esa capacidad a nivel local. Allí, las muestras pudieron ser secuenciadas por primera vez a principios de junio pasado y la presencia de dos de las variantes de preocupación se confirmó.
La capacidad para hacer pruebas de PCR, un paso previo y necesario a la secuenciación, también fue fortalecida en Haití a través de la cooperación de la OPS.
"Recibimos apoyo técnico, capacitación, insumos, reactivos y apoyo financiero de la OPS. Desde 2015, la OPS nos ha apoyado con dos técnicos de laboratorio para hacer la vigilancia de otros virus. Ahora, desde el comienzo de la pandemia, nos están apoyando con otros dos técnicos en mi laboratorio para hacer las pruebas más rápidamente”.
Ito Journel, coordinador responsable por el servicio de biología molecular del Laboratorio Nacional de Salud Pública de Haití.
Con los insumos disponibles, el país puede hacer la vigilancia y tamizaje inicial de tres de las variantes de preocupación. Y por su participación en la red COVIGEN, puede acceder a los laboratorios de referencia para secuenciar muestras previamente seleccionadas bajo criterios establecidos, para confirmar o detectar variantes de preocupación, incluida delta.
“Ahora queremos centrarnos en saber si la variante delta ha ingresado en el país. Sabemos que hay algunos casos en República Dominicana y en la Florida, y que por la gente que viaja hacia Haití, es posible que la tengamos, pero para confirmarlo tenemos que enviar muestras para secuenciar”, indica sobre un procedimiento que comenzarán a hacer cada mes.
El técnico de laboratorio haitiano ha obtenido una beca para hacer un máster en Fiocruz, algo que le permitirá implementar la tecnología para hacer secuenciación en Haití cuando finalice sus estudios en dos años.
Considera que tener la posibilidad de hacer secuenciación es muy importante, especialmente en una pandemia como ésta, cuando los laboratorios de referencia pueden estar sobrecargados para hacer pruebas para otros países. Tener esa capacidad instalada en el país también es una oportunidad para expandir la vigilancia genómica para otros virus. “Sería importante tener la capacidad de hacer esta vigilancia genómica en el país. No es solo para SARS-CoV-2, sino para cualquier otro virus que necesitemos hacer la secuencia”, sostiene.
Mientras Ito realiza su maestría en Brasil, algo que comenzará en la segunda quincena de agosto de 2021, un equipo continuará con el trabajo liderado por él en Haití. Pero Ito asegura que, por email o mensajería instantánea, seguirá siempre en contacto con su laboratorio e involucrado con la vigilancia del SARS-CoV-2 en su país.
Honduras: Red de Vigilancia Genómica ayuda a confirmar la variante alpha
En junio de 2021, el Laboratorio Nacional de Virología de Honduras analizó muestras de pacientes positivos con COVID-19 y detectó a través de la prueba PCR la presencia de algunas de las variantes de preocupación. Sin embargo, y al carecer de la capacidad para hacer secuenciación y confirmar la identidad de esas muestras o de encontrar otras variantes que escapan a la PCR de tamizaje, la doctora Mitzi Castro recurrió al Instituto Gorgas de Panamá, uno de los seis laboratorios de referencia de la Red Regional de Vigilancia Genómica (COVIGEN). Con el apoyo de la OPS, las muestras llegaron a Panamá y días después, el Gorgas confirmó la presencia de la variante de preocupación alpha en Honduras.
“Ahora el país está actualizado en los datos sobre las variantes y eso tiene relevancia porque permite a los médicos y a las autoridades sanitarias tomar acciones que conduzcan a enfrentar las variantes en el país”, afirma Castro, directora del Laboratorio Nacional de Virología de la Secretaría de Salud de Honduras.
A partir del apoyo de la OPS, el laboratorio hondureño puedo realizar las pruebas PCR para detección de las variantes alpha, beta y gamma, y hacer así vigilancia de estos virus en el país. “Ahora tenemos el protocolo para VOC (variantes de preocupación) que permite tamizar tres de las cuatro variantes de preocupación que comparten las mismas mutaciones, y funciona muy bien”, cuenta.
Sin embargo, el tamizaje no reemplaza la secuenciación y los protocolos y PCR que permiten detectar las variantes alpha, beta y gamma, no funcionan para la variante delta, ya que no posee la misma mutación. Por lo tanto, “el país incluye en los envíos a los laboratorios de referencia muestras con resultados negativos para las tres variantes con el fin de identificar delta o cualquier otra variante por secuenciación”, algo que sigue las recomendaciones de la OPS.
Mitzi Castro destaca el apoyo de la OPS en la elaboración de los criterios para la selección de muestras a secuenciar, en el envío de estas a los laboratorios de la Red COVIGEN, en la entrega de reactivos para hacer las pruebas de PCR a nivel local, así como en la capacitación para compartir los resultados a nivel global a través de la plataforma GISAID.
La jefa del laboratorio dice que los análisis del laboratorio han permitido arrojar un dato que considera importante: “estamos teniendo gente joven, de 15 a 48 años, más afectada por las variantes, y estamos dando a conocer esto a través de los medios, con información adecuada y correcta, para que la población lo conozca y tome acción, cambie su comportamiento y se proteja”.
En Honduras circulan también otras variantes comunes, pero hasta ahora no se ha confirmado circulación de la variante delta.
Castro asegura que, tras la primera confirmación de la presencia de otras variantes en el país, continuarán “con la vigilancia todas las semanas, haciendo protocolos para tamizaje de variantes, recolectando muestras y analizando los datos, y enviando muestras a los laboratorios de referencia para secuenciar y detectar la presencia de cualquier otra variante de preocupación”. Como reflexión final, la especialista subraya que: “el laboratorio es el pilar del diagnóstico y los resultados oportunos permiten tomar acciones oportunas”.
¿Qué es la secuenciación genética?
El virus SARS-CoV-2, así como otros virus como los de la influenza, cambian constantemente; sufren cambios genéticos con el tiempo y la alta circulación, cambios en el genoma del virus, algo conocido como “mutaciones” o “variantes”.
La secuenciación genética es una técnica de laboratorio que permite leer el genoma de un virus. Con este método se pueden descubrir nuevos patógenos, como ha sucedido con el SARSCoV-2, ya que cada organismo posee una secuencia genómica exclusiva.
El estudio de la evolución del SARS-CoV-2 sirve para la detección de mutaciones o variantes que puedan modificar la tasa de transmisión o el poder patógeno del virus o mermar la utilidad de las medidas de respuesta médica, como las pruebas diagnósticas, vacunas y tratamientos.
Asimismo, el seguimiento geográfico y temporal de las mutaciones del virus puede ayudar a rastrear su propagación y a conocer mejor las posibles vías y la dinámica de transmisión.
Así, la secuenciación genómica permite monitorear los cambios que sufre el genoma del virus, recopilar información sobre esas mutaciones, para ayudar a determinar si el virus se ha vuelto más transmisible, si las vacunas existentes siguen siendo efectivas o si las pruebas de diagnóstico aún detectan el virus con sus cambios, entre otras.
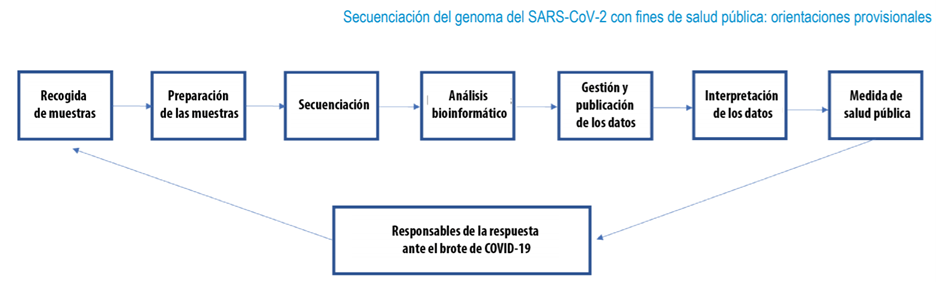
De la toma de muestras a las medidas de salud pública
El trabajo para conocer las variantes que circulan en un país comienza con la recogida de muestras. Por ejemplo, una persona con síntomas que fue a un servicio de salud y le hicieron la prueba de PCR y su resultado dio positivo para COVID-19.
Esa y otras muestras representativas del país, son enviadas al laboratorio nacional de virología de un país donde se preparan las muestras para la secuenciación. Esto implica la selección del material que pasará a ser secuenciado.
Esa selección debe basarse en criterios definidos no solo de calidad, sino además para asegurar una buena representación de las variantes circulando en el país. Previo a la secuenciación, el laboratorio de salud pública nacional puede realizar una serie de ensayos que funcionan igual al diagnóstico, diseñados para reconocer algunas de las mutaciones del virus y tamizar entre las muestras para ver cuáles pueden ser alpha, beta o gamma, o incluso algunos ensayos que detectan cambios específicos de cada variante.
Sin embargo, estos ensayos son considerados preliminares y deben ser complementados con la secuenciación.
Aunque algunos SARS-CoV-2 circulantes pueden presentar mutaciones específicas asociadas a estas variantes, los ensayos de tamizaje por sí solos no son suficientes para clasificar el virus como una de las variantes de preocupación o de interés reconocidas. En este sentido, la secuenciación genómica completa es requerida para que se pueda confirmar la variante de preocupación”, explica Juliana Leite, especialista en laboratorio y enfermedades virales de la OPS.
Tras la secuenciación, el personal de laboratorio pasa a realizar un análisis bioinformático. Esto permite confirmar la presencia de alpha, beta o gamma, identificar delta y cualquier otra variante que pueda surgir. Este análisis es parte de la rutina de vigilancia genómica y es realizado por los países que tienen capacidad para hacerlo o pueden ser apoyados por alguno de los laboratorios de referencia de la Red Regional de Vigilancia Genómica.
Finalizado el análisis, los datos son publicados en GISAID para que la información esté disponible a nivel global para cualquiera que la requiera. La amplia difusión de las secuencias de SARS-CoV-2 y de los procedimientos diagnósticos y los protocolos y las muestras de secuenciación ha posibilitado el desarrollo de capacidad de vigilancia genómica del virus SARS-CoV-2 en todo el mundo.
Así, por medio de la vigilancia genómica del SARS-CoV-2, los expertos interpretan los datos obtenidos para conocer las linajes y variantes circulantes, así como sus mutaciones y luego comparten su análisis con los responsables de la respuesta a la pandemia para asesorar las medidas de salud pública y las recomendaciones a la población.