A estrutura da rede inclui uma combinação de países com capacidade de sequenciamento interno e aqueles que enviam para sequenciamento externo a um dos dois Laboratórios Regionais de Sequenciamento (Fundação Oswaldo Cruz/FIOCRUZ - Brasil e Instituto de Saúde Pública/ISPCH-Chile). Além disso, treinamentos regionais e nacionais e ações adicionais de suporte para gerar informações oportunas sobre dados de sequenciamento genômico do SARS-CoV-2 estão disponíveis por meio da rede.
COV|GEN
Rede Regional de Vigilância Genômica de COVID-19
A OPAS incentiva os laboratórios a sequenciar amostras positivas de COVID-19 e compartilhar informações genéticas oportunamente através da plataforma GISAID (do inglês, Global Initiative on Sharing All Influenza Data).
A participação na Rede Regional de Vigilância Genômica COVID-19 é aberta a todos os países das Américas por meio dos Laboratórios Nacionais de Saúde Pública. Para mais informações, entre em contato com o Escritório Regional da OPAS por e-mail leitejul@paho.org, ricoj@paho.org.
Laboratórios de Sequenciamento Genômico de SARS-CoV-2
© Organização Pan-Americana da Saúde, 2020. Todos os direitos reservados.
Produção de relatórios: Departamento de Emergências de Saúde da OPAS (PHE): Equipe de Vigilância da Influenza / Gestão de Riscos Infecciosos (IHM) e Gestão de Dados, Análise e Produtos / Informações sobre Emergências de Saúde e Avaliações de Riscos (HIM).
Isenção de responsabilidade: As designações utilizadas e a apresentação do material nestes mapas não implicam a expressão de qualquer opinião por parte da Secretaria da Organização Pan-Americana da Saúde sobre a situação jurídica de qualquer país, território, cidade ou zona ou de suas autoridades. , ou na delimitação de suas fronteiras ou limites. As linhas pontilhadas e tracejadas nos mapas representam as linhas de fronteira aproximadas para as quais pode não haver acordo total.
Variantes de SARS-CoV-2 nas Américas
Desde a caracterização genômica inicial do SARS-CoV-2, o vírus evoluiu em diferentes grupos genéticos. A ocorrência de mutações é um evento natural e esperado dentro do processo de evolução do vírus.
- Mutação refere-se a qualquer mudança no genoma viral.
- Algumas mutações específicas definem os grupos genéticos virais que circulam atualmente mundialmente.
- Em geral, Um vírus mutado é chamado de variante do vírus original.
- Existem diferentes tipos de mutações. As variantes podem diferir em uma ou mais delas.
- Apesar da maiorai das mutações não ter nenhum impacto, algumas podem fazer com que o vírus seja mais transmissível, ou facilitar o escape do vuris da resposta imune.
- Na comunidade, quanto mais elevado é o nível de transmissão, maior é a probabilidade de que ocorra mutações no virus.
Quando essas variantes têm um potencial impacto ou risco para a saúde pública, elas são consideradas variantes de preocupação (VOC, do inglês Variant of Concern)
Classificação de Variantes1
Um isolado de SARS-CoV-2 é uma variante de interesse (VOI) se muda fenotipicamente em comparação com um isolado de referência ou tiver um genoma com mutações que levem a alterações de aminoácidos associadas a implicações fenotípicas estabelecidas ou suspeitas;
E
foi identificado que causar transmissão comunitária/múltiplos casos/clusters COVID-19, ou foi detectado em vários países;
OU
foi avaliado como uma VOI pela OMS em consulta com o Grupo de Trabalho de Evolução do Vírus SARS-CoV-2 da OMS.
Uma VOI é uma variante de preocupação (VOC) se, através de uma avaliação comparativa, tiver demostrado que está associada a
- Aumento da transmissibilidade ou alteração prejudicial na epidemiologia da COVID-19;
- Aumento da virulência ou mudança na apresentação clínica da doeça; ou
- Diminuição da eficácia das medidas sociais e de saúde pública ou do diagnósticos, vacinas, terapias disponíveis.
OU
foi avaliado como uma VOC pela OMS em consulta ao Grupo de Trabalho de Evolução do Vírus SARS-CoV-2 da OMS
1https://www.who.int/publications/m/item/covid-19-weekly-epidemiological-update
GISAID - Sistema de rastreamento de variantes
Monitoramento de variantes SARS-CoV-2
Mutações ou variantes virais estão sendo monitoradas desde o início da pandemia da COVID-19 através do banco de dados de sequencia GISAID (do inglês, Global Initiative on Sharing Avian Influenza Data). A OMS avalia rotineiramente se as variantes de SARSCoV-2 têm impacto sobre:
- Transmissibilidade do vírus
- Gravidade da doença
- Efetividade de diagnósticos, terapias e vacinas
- Avaliação de risco das variantes de preocupação para determinar se haverá implicações para a saúde pública é realizada rotineiramente.
Principais ações de um Estado-Membro, se uma VOI ou VOC for identificada:
- Reportar imediatamente à OPAS/OMS casos/conglomerados iniciais associados à infecção por VOI ou VOC através do mecanismo do Regulamento Sanitário Internacional (RSI).
- Enviar sequências completas do genoma e metadados associados a um banco de dados disponível publicamente, como o GISAID.
- Quando existir capacidade e em coordenação com a comunidade internacional, realizar investigações de campo para melhorar a compreensão dos potenciais impactos do VOI ou VOC na epidemiologia COVID-19, gravidade, eficácia da saúde pública e medidas sociais, ou outras características relevantes.
- Realizar avaliações laboratoriais ou entrar em contato com a OPAS para obter suporte para realizar avaliações laboratoriais sobre o impacto da VOI ou VOC em métodos de diagnósticos, respostas imunes, neutralização de anticorpos ou outras características relevantes.
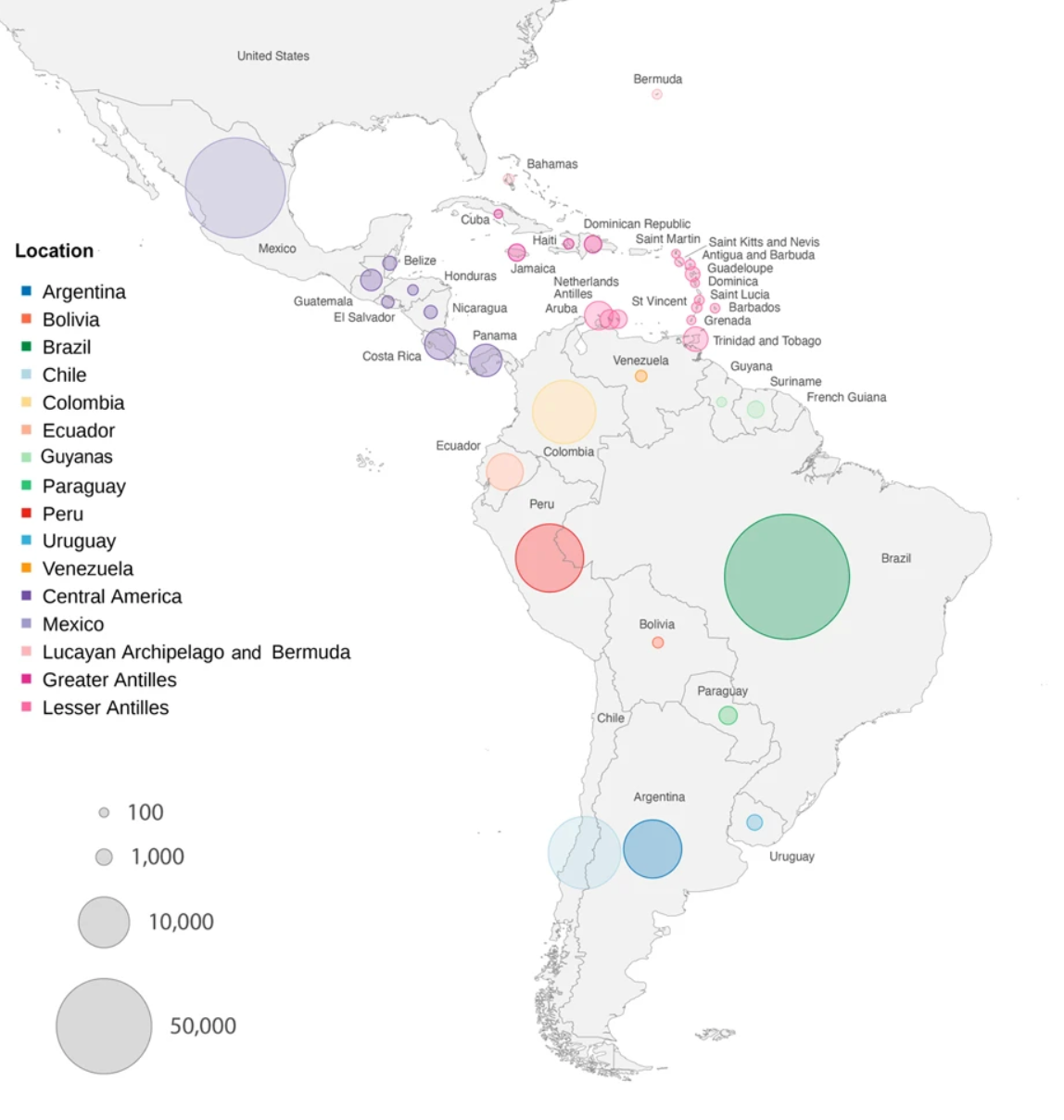
Dispersion patterns of SARS-CoV-2 variants Gamma, Lambda and Mu in Latin America and the Caribbean
Published: 28 February 2024 - Nature Communications
Latin America and Caribbean (LAC) regions were an important epicenter of the COVID-19 pandemic and SARS-CoV-2 evolution. Through the COVID-19 Genomic Surveillance Regional Network (COVIGEN), LAC countries produced an important number of genomic sequencing data that made possible an enhanced SARS-CoV-2 genomic surveillance capacity in the Americas, paving the way for characterization of emerging variants and helping to guide the public health response. In this study we analyzed approximately 300,000 SARS-CoV-2 sequences generated between February 2020 and March 2022 by multiple genomic surveillance efforts in LAC and reconstructed the diffusion patterns of the main variants of concern (VOCs) and of interest (VOIs) possibly originated in the Region. Our phylogenetic analysis revealed that the spread of variants Gamma, Lambda and Mu reflects human mobility patterns due to variations of international air passenger transportation and gradual lifting of social distance measures previously implemented in countries. Our results highlight the potential of genetic data to reconstruct viral spread and unveil preferential routes of viral migrations that are shaped by human mobility patterns.