La estructura de la Red incluye una combinación de países con capacidad de secuenciación internamente, y aquellos que requieren el envío para secuenciación externa a cualquiera de los 8 Laboratorios Regionales de Secuenciación, que están en capacidad de proporcionan secuenciación externa a los laboratorios participantes. Además, capacitaciones regionales y a nivel de país, y acciones de apoyo adicionales para generar información oportuna de los datos de secuenciación genómica de SARS-CoV-2 están disponibles también a través de la Red.
Laboratorios Regionales de Secuenciación: Fundação Oswaldo Cruz – Brasil; Instituto de Salud Pública de Chile – Chile; Instituto Nacional de Salud – Colombia; Instituto Costarricense de Investigación y Enseñanza en Nutrición y Salud – Costa Rica; Instituto de Diagnóstico y Referencia Epidemiológicos – México; Instituto Conmemorativo Gorgas de Estudios de la Salud- Panamá; University of the West Indies - Trinidad y Tobago; Centers for Disease Control and Prevention – Estados Unidos de la America.
COV|GEN
Red Regional de Vigilancia Genómica de COVID-19
La OPS alienta a los laboratorios a secuenciar muestras positivas COVID-19 y compartir oportunamente información genética a través de la plataforma GISAID (del inglés, Global Initiative on Sharing All Influenza Data).
La participación en la Red Regional de Vigilancia Genómica COVID-19 está abierta a todos los países de las Américas a través de los Laboratorios Nacionales de Salud Pública. Para obtener información adicional, por favor entrar en contacto con la Oficina Regional de la OPS a través de los correos electrónicos leitejul@paho.org, ricoj@paho.org.
Laboratorios de secuenciación genómica de SARS-CoV-2
© Organización Panamericana de la Salud, 2020. Todos los derechos reservados.
Producción de informes: Departamento de Emergencias Sanitarias (PHE) de la OPS: Equipo de Vigilancia de Influenza / Gestión de Riesgos Infecciosos (IHM) y Gestión de Datos, Análisis y Productos / Información de Emergencias Sanitarias y Evaluaciones de Riesgos (HIM).
Descargo de responsabilidad: Las designaciones empleadas y la presentación del material en estos mapas no implican la expresión de opinión alguna por parte de la Secretaría de la Organización Panamericana de la Salud sobre la situación jurídica de ningún país, territorio, ciudad o zona o de sus autoridades. , o en la delimitación de sus fronteras o límites. Las líneas punteadas y discontinuas en los mapas representan líneas fronterizas aproximadas para las cuales puede que aún no haya un acuerdo total.
Variantes de SARS-CoV-2 en las Américas
Desde la caracterización genómica inicial del SARS-CoV-2, el virus ha evolucionado en diferentes grupos genéticos. La ocurrencia de mutaciones es un evento natural y esperado dentro del proceso de evolución del virus.
- Mutación se refiere a cualquier cambio en el genoma del virus.
- Algunas mutaciones específicas definen los grupos genéticos virales que circulan actualmente en todo el mundo.
- En general, un virus mutado se considera variante del virus original.
- Existen diferentes tipos de mutaciones. Las variantes pueden diferir en una o más de ellas.
- Aunque la mayoria de mutaciones no tiene ningun impacto, algunas pueden resultar en que el virus sea más transmisible, o facilitar que el virus se escape de la respuesta inmune.
- En la comunidad, cuanto mas elevado es el nivel de transmision, mayor es la probabilidad de que ocurran mutaciones en el virus.
Cuando estas variantes tienen un potencial impacto o riesgo para la salud publica, se consideran variantes de preocupación (VOC, del inglés Variant of Concern)
Clasificación de Variantes1
Un aislado de SARS-CoV-2 es una variante de interés (VOI) si cambia fenotípicamente en comparación con un aislado de referencia, o tiene un genoma con mutaciones que conducen a cambios de aminoácidos asociados con implicaciones fenotípicas establecidas o sospechosas;
Y
se ha identificado que causa transmisión comunitaria/múltiples casos/clústeres de COVID-19, o se ha detectado en varios países;
O
fue evaluado como una VOI por la OMS en consulta con el Grupo de Trabajo sobre la Evolución del Virus SARS-CoV-2 de la OMS.
Una VOI es una variante de preocupación (VOC) si, a través de una evaluación comparativa, se ha demostrado que está asociada con
- Aumento de la transmisibilidad o cambio perjudicial en la epidemiología de la COVID-19;
- Aumento de la virulencia o cambio en la presentación clínica de la enfermedad; o
- Disminución de la eficacia de las medidas sociales y de salud pública o del diagnóstico disponible, vacunas, terapias.
O
fue evaluado como una VOC por la OMS en consulta con el Grupo de Trabajo sobre la Evolución del Virus SARS-CoV-2 de la OMS
1https://www.who.int/publications/m/item/covid-19-weekly-epidemiological-update
GISAID - Seguimiento de VARIANTES
Monitoreo de variantes SARS-CoV-2
Mutaciones o variantes virales han sido monitoreadas desde el inicio da pandemia da COVID-19 a través del banco de datos de secuencia GISAID (del inglés, Global Initiative on Sharing Avian Influenza Data). La OMS evalúa rutineramente si las variantes de SARS-CoV-2 tienen impacto sobre:
- Transmisibilidad del virus
- Gravedad de la enfermedad
- Eficacia de los diagnósticos, terapias y vacunas
- Risk assessment for variants of concern to determine if there will be public health implications are routinely performed.
Acciones principales de un Estado miembro, si se identifica una VOI o VOC:
- Informar inmediatamente a la OPS/OMS casos/conglomerados iniciales asociados con la infección por VOI o VOC a través del mecanismo del Reglamento Sanitario Internacional (RSI)
- Enviar secuencias completas del genoma y metadatos asociados a una base de datos disponible públicamente, como GISAID.
- Cuando exista capacidad y en coordinación con la comunidad internacional, realizar investigaciones en terreno para mejorar la comprensión de los posibles impactos del VOI o VOC en la epidemiología de la COVID-19, gravedad, eficacia de la salud pública y medidas sociales u otras características pertinentes.
- Realizar evaluaciones de laboratorio o ponerse en contacto con la OPS para obtener apoyo para realizar evaluaciones de laboratorio sobre el impacto de la VOI o VOC en métodos de diagnóstico, respuesta inmune, neutralización de anticuerpos u otras características pertinentes
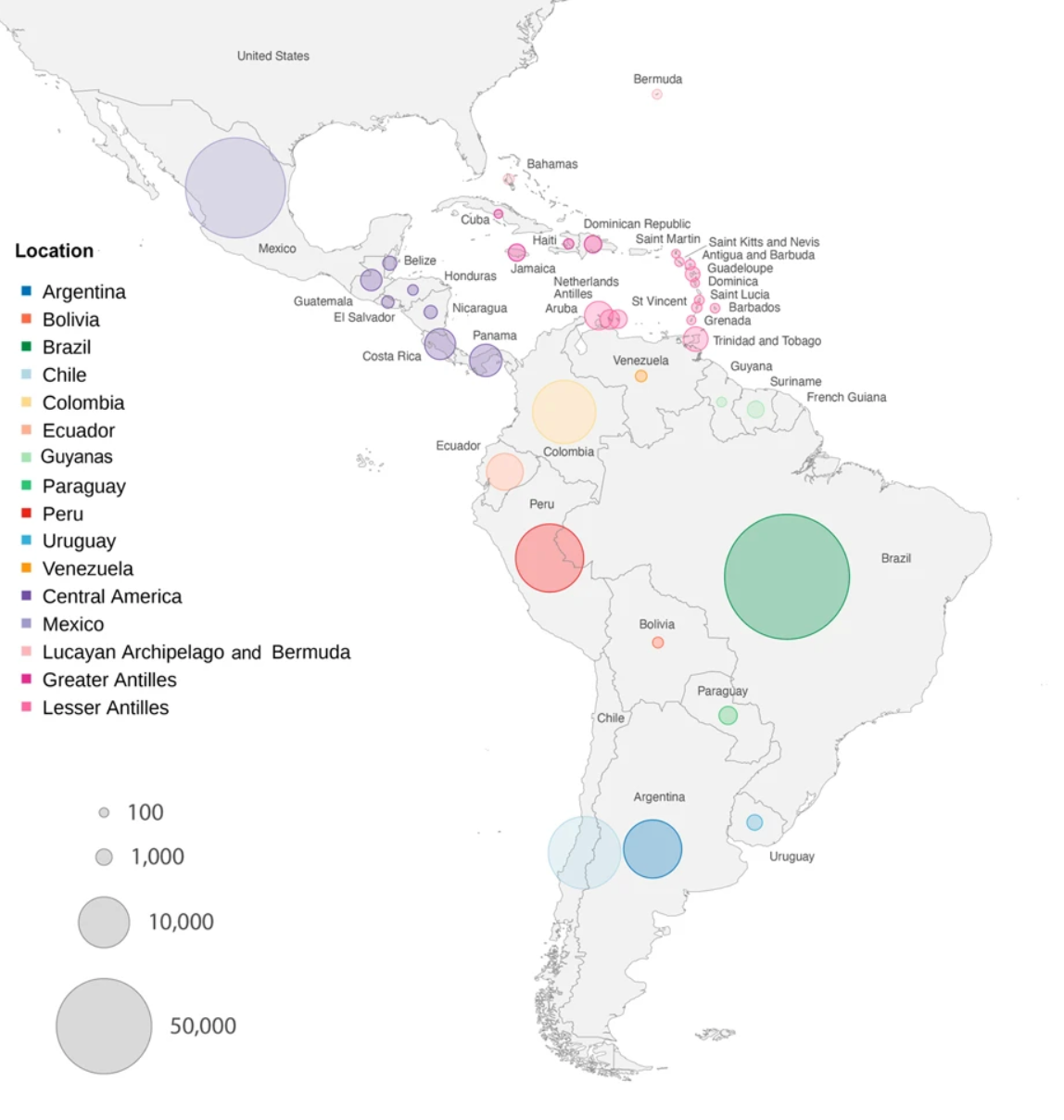
Dispersion patterns of SARS-CoV-2 variants Gamma, Lambda and Mu in Latin America and the Caribbean
Published: 28 February 2024 - Nature Communications
Latin America and Caribbean (LAC) regions were an important epicenter of the COVID-19 pandemic and SARS-CoV-2 evolution. Through the COVID-19 Genomic Surveillance Regional Network (COVIGEN), LAC countries produced an important number of genomic sequencing data that made possible an enhanced SARS-CoV-2 genomic surveillance capacity in the Americas, paving the way for characterization of emerging variants and helping to guide the public health response. In this study we analyzed approximately 300,000 SARS-CoV-2 sequences generated between February 2020 and March 2022 by multiple genomic surveillance efforts in LAC and reconstructed the diffusion patterns of the main variants of concern (VOCs) and of interest (VOIs) possibly originated in the Region. Our phylogenetic analysis revealed that the spread of variants Gamma, Lambda and Mu reflects human mobility patterns due to variations of international air passenger transportation and gradual lifting of social distance measures previously implemented in countries. Our results highlight the potential of genetic data to reconstruct viral spread and unveil preferential routes of viral migrations that are shaped by human mobility patterns.